Le jumeau numérique, allié des patients et des médecins
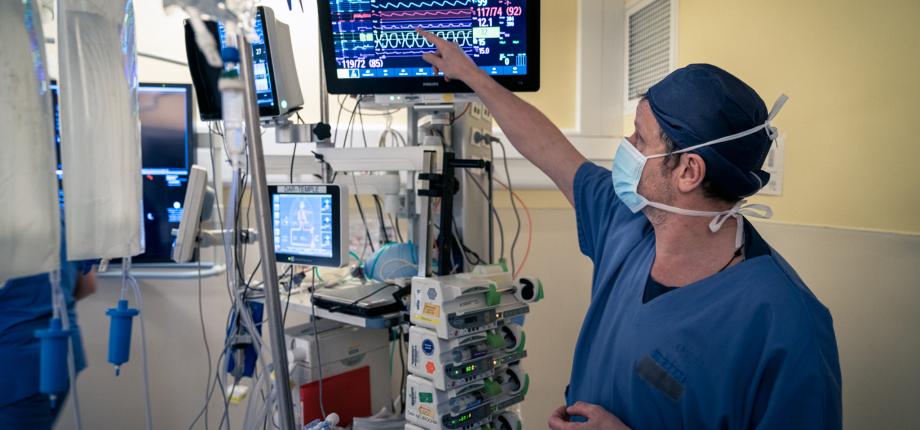
Au bloc opératoire, un médecin anesthésiste vous endort. Tout au long de l’intervention, il surveillera vos constantes et les maintiendra en fonction des informations qu’il a sur vous. Mais aujourd’hui, vous n’êtes pas le seul patient en salle d’opération. Votre jumeau numérique vous accompagne. « Le jumeau numérique est un logiciel dans lequel des éléments de modélisation concernant le patient sont compilés. Il sait prédire des effets à partir d’une cause et aide le médecin anesthésiste à anticiper ses gestes », indique Dominique Chapelle, directeur de recherche Inria et membre de l’équipe de recherche Inria – École polytechnique M3DISIM.
Le praticien surveille notamment votre tension artérielle afin de garantir la bonne irrigation sanguine de vos organes. Lorsque celle-ci chute (hypoperfusion), il réagit et réalise une suite d’actes pour rétablir la situation. Cela peut demander du temps et, dans certains cas, laisser des séquelles (cognitives, cardiaques…). « Le jumeau numérique lui, est en mode prédictif. L’algorithme traite les données qu’il connait déjà de vous et agrège en parallèle de la data sur le déroulé de l’opération, produisant ainsi de l’information supplémentaire utile au monitorage de l’anesthésie. Le médecin peut l’interroger à tout moment et avoir une vision sur les dérives possibles dans les 2 ou 3 prochaines minutes afin d’adapter au mieux ses actes et de gagner en efficacité », ajoute le chercheur. De telles fonctions sont aussi précieuses en réanimation où les patients sont intubés et placés en coma artificiel (l’équivalent d’une anesthésie générale prolongée). Le jumeau numérique est alors apte à faire des prédictions sur l’évolution de leur maladie, permettant aux soignants de réaliser des bilans et d’adapter les soins en limitant la multiplication de gestes invasifs.
Avec l’aide de l’intelligence artificielle
Pour parvenir à un tel résultat, l’équipe M3DISIM développe des jumeaux numériques basés sur une modélisation dite mécanistique. « Il s’agit de décrire par des équations des relations de causes à effets, en disséquant un maximum de phénomènes. Cela demande d’agréger des données sur chaque cause et chaque effet impliqué. En biologie par exemple, il faut pouvoir observer et mesurer les mécanismes en jeu au niveau cellulaire. C’est complexe et chronophage mais nous nous aidons parfois de l’intelligence artificielle pour alléger cette difficulté », explique Dominique Chapelle.
Les algorithmes d’apprentissage automatique abordent en effet cet enchainement de causes et d’effets comme une boîte noire dans laquelle la mécanique en jeu n’a pas été décortiquée et mise en équation. Il est alors possible d’entrainer des réseaux de neurones en basant leur travail sur les données connues en entrée et en sortie de la boîte. La méthode est par exemple utile en anesthésie pour déterminer les effets de certains médicaments à l’échelle des cellules cardiaques, sans disposer de toutes les données afférentes. En croisant les informations personnelles et fonctionnelles du patient (paramètres cardiaques, manière dont le cœur travaille, pathologie, etc.), la concentration du produit injecté et ses effets connus, l’intelligence artificielle prédit les effets pharmacologiques de la drogue utilisée.
Par ailleurs, « un modèle mécanistique qui rencontre dans les données personnelles et fonctionnelle d’un patient les caractéristiques d’une pathologie dont on a mis en équation les causes et les effets, est capable d’élaborer des prédictions cohérentes en travaillant avec cette pathologie », souligne Dominique Chapelle. C’est un avantage considérable sur les modèles statistiques, ces derniers ne pouvant en effet faire des prédictions que sur des données relatives à des situations déjà rencontrées. Les modèles mécanistiques présentent également l’avantage, par les relations de causes à effets qu’ils intègrent, de fournir des prédictions parfaitement analysables et donc explicables, ce qui en facilite aussi l’acceptabilité.
Appréhender le vivant
Aujourd’hui, les applications des travaux de Dominique Chapelle se concentrent sur la médecine d’anesthésie et de réanimation, en partenariat avec l’hôpital Lariboisière à Paris et l’Inserm (laboratoire MASCOT). De précédentes recherches l’ont toutefois conduit à établir des jumeaux numériques de cœur dans le but d’optimiser l’implantation de pacemaker en fonction des particularités des patients. Ces outils sont néanmoins applicables à tous les organes. Martin Genet, également membre de l’équipe M3DISIM et chercheur au LMS mène ainsi des recherches sur les jumeaux numériques de poumons afin de mieux comprendre certaines maladies chroniques comme la fibrose pulmonaire idiopathique. « Il faut alors pouvoir valider nos modèles en vérifiant qu’ils reproduisent bien la relation de cause à effet que l’on a cherché à décrire. Cela nécessite de nombreuses données qu’il n’est pas toujours facile d’obtenir car le vivant ne se laisse pas appréhender facilement. C’est un véritable challenge », conclut Dominique Chapelle.

A propos
Dominique Chapelle est directeur de recherche Inria, chercheur au Laboratoire de Mécanique des Solides de l’École polytechnique et membre de l’équipe de recherche Inria – École polytechnique M3DISIM. Il est également responsable scientifique du laboratoire commun AP-HP - Inria Daniel Bernoulli (Bernoulli Lab).
Ses travaux portent notamment sur la modélisation biomécanique et notamment celle du système cardiovasculaire. Il croit fondamentalement à l’utilité des modèles mécanistiques et est fasciné par l’idée de comprendre le vivant à diverses échelles.
Ses autres recherches incluent les méthodes numériques et l’analyse mathématique pour la mécanique des structures, mais aussi les problèmes inverses associés.
*LMS : une unité mixte de recherche CNRS, École polytechnique, Institut Polytechnique de Paris, 91120 Palaiseau, France













